为什么写这篇文章?
这是我在为完成《蛋白质工程》课程展示任务过程中写的,选题自然是cas蛋白,我发现在网络和各类博客上反而很难找到cas9蛋白的具体内容,它被介绍CRISPR-Cas的文章淹没了,在读了十几篇文献后,用latex写了笔记。鉴于关于CRISPR-Cas的内容随处可见,完全可以去看产品手册或者其他什么,这篇文章中删去了与Cas蛋白无直接关系的大量内容。
Cas蛋白简介
Cas是CRISPR相关蛋白的简称, 簇状规则间隔的短回文重复序列(CRISPR)和相关蛋白(Cas)构成CRISPR–Cas系统1,是许多细菌和大多数古细菌中存在的抗噬菌体免疫系统。 近年来,CRISPR–Cas系统已发展成为可靠而强大的基因组编辑工具。

第2类CRISPR-Cas系统的特征是由单一、大、多结构域的蛋白质组成复合体发挥作用,一些2类效应蛋白,如Cas9,已经成功地用于基因组工程。3


Cas9蛋白结构
Cas9蛋白立体结构


Cas9蛋白结构域


Cas9蛋白功能及作用机制
Cas9蛋白在CRISPR-Cas系统中的功能
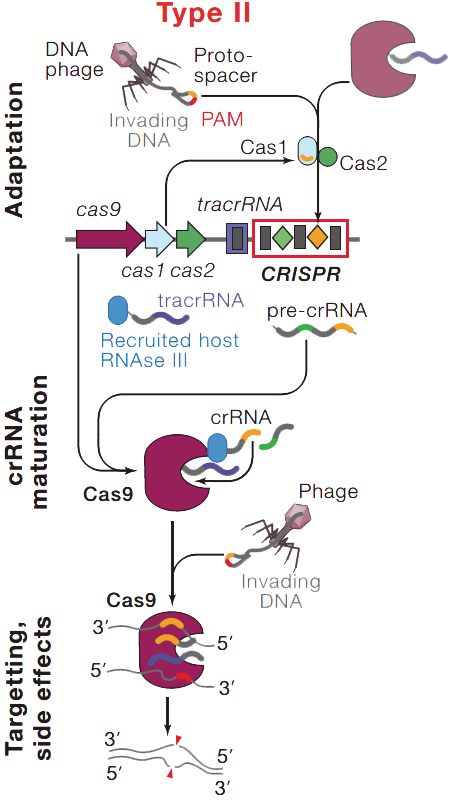
上图8显示的是Type II 系统的作用过程。3 首先是依靠Cas1、2复合体将原间隔序列的片段制作成间隔序列整合进CRISPR序列中,形成对外源DNA的记忆,然后在接下来的免疫中,间隔序列就能快速产生原间隔序列片段,这些序列片段与Cas9一起靶向地切除在细菌内的外源DNA,PAM在这里参与防止自身免疫疾病的机制,保障不会切除自身DNA。
Cas9蛋白功能的机制
结合DNA并识别目标序列

就这样借助sgRNA的部分序列与靶DNA位点进行碱基配对,能够引导Cas9结合到这个靶位点上并进行切割。II型和V-B型系统需要tracrRNA才能正常发挥功能,而V-A型系统则需要单独使用crRNA 。在实际应用时,人们可以将tracrRNA和crRNA作为两种向导RNA(gRNA)或者融合在一起形成单向导RNA(single guide RNA, sgRNA),后一种方式已经广泛用于Cas9引导酶Cas9结合到靶DNA序列上并进行切割,Cas9与sgRNA一起被称作Cas9-sgRNA系统。


残基Glu 1108和ser 1109与靶DNA链中的+1磷酸二酯基团相互作用,直接导致PAM上游的局部链分离,将靶链转为与sgRNA结合(1-2bp),靶链DNA与sgRNA之间的碱基配对促进了引导-靶标异质双链的进一步逐步置换和传播。7
切割
Cas9的基因组编辑能力只有在被称作前间隔序列邻近基序(protospacer adjacent motif, PAM)的短片段DNA序列的存在下才成为可能。只有DNA靶位点附近存在PAM时,Cas9才能进行准确切割。PAM的存在也是激活酶Cas9所必需的,PAM 序列的特征为 NGG(其中 N 为任意核苷酸)。8
核酸酶Cas9含有两个具有切割活性的结构域:HNH结构域和RuvC结构域,其中 HNH结构域切割与crRNA互补的DNA链,而RuvC结构域切割非互补链(即HNH和 RuvC结构域协同切割TS和NTS)。RuvC结构域可分为三个亚结构域:RuvC I、RuvC II和RuvC III,RuvC I接近于Cas9的氨基端,RuvC II和RuvC III位于HNH结构域的两侧(见图5)。9
当Cas9与靶基因位点结合时发生了构象变化,核酸酶功能区对靶标DNA的反向链进行定位切割。Cas9介导的DNA切割最后结果是目标DNA(PAM序列上游约
3~4个核苷酸)的双链断裂(double strand break,DSB)。

传统的Cas9蛋白包含RuvC和HNH两个催化结构, RuvC和HNH可分别剪切DNA的两条链形成双链断裂,如gRNA与非目标区域结合就有造成不必要插入突变的可能。
其他
存在的问题
基因组极为复杂,gRNA可能与非靶向序列局部匹配,这种局部匹配也会激活Cas 9内切酶活性,从而产生脱靶效应。11
此外,Cas9不仅识别标准PAM,也可识别非标准PAM,这也可能会引起一定程度的脱靶。脱靶可能影响正常基因的功能表达,甚至激活致癌因子、抑制抑癌基因,造成安全隐患,这极大的阻碍了该技术在临床的进一步应用。目前主要通过优化gRNA 设计,改造Cas9蛋白,使用RNP递送方式等策略来提高CRISPR/Cas9系统特异性以降低脱靶现象。
Cas9蛋白突变体
博德研究所的研究人员对Cas9进行突变使RuvC和HNH两个催化结构域中的一个缺失核酸酶活性形成Cas9n,Cas9n与DNA双链作用时仅产生单链切口。应用CRISPR/Cas9n系统进行基因编辑需要使用两个相邻且相反链上的gRNA序列。虽然所用的每条gRNA的脱靶结合位点可能出现在全基因组范围内,但是Cas9n仅催化每个位置的单链断裂 (Single-strand break,SSB)。SSB优先通过HDR进行修复,而不是NHEJ,这可降低不必要的插入缺失突变的发生。应用CRISPR/Cas9n系统进行基因编辑能将脱靶活性降低50-1000倍。12
-
本文不介绍与Cas蛋白无关的CRISPR–Cas技术细节,有兴趣的详见 CRISPR Guide ↩︎
-
Makarova K S , Haft D H , Barrangou R , et al. Evolution and classification of the CRISPR–Cas systems[J]. Nature Reviews Microbiology. ↩︎
-
Makarova K S , Zhang F , Koonin E V . SnapShot: Class 2 CRISPR-Cas Systems[J]. Cell, 2017, 168(1-2):328-328. ↩︎
-
DOI: 10.2210/pdb6VPC/pdb ↩︎
-
Anders, Carolin, Niewoehner, et al. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease.[J]. Nature, 2014. ↩︎
-
Lapinaite A , Knott G J , Palumbo C M , et al. DNA capture by a CRISPR-Cas9–guided adenine base editor[J]. ence, 2020, 369(6503):566-571. ↩︎
-
Anders, Carolin, Niewoehner, et al. Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease.[J]. Nature, 2014. ↩︎
-
Nierzwicki U , Arantes P R , Saha A , et al. Establishing the allosteric mechanism in CRISPR‐Cas9[J]. Wiley Interdiplinary Reviews: Computational Molecular ence. ↩︎
-
Huai C , Li G , Yao R , et al. Structural insights into DNA cleavage activation of CRISPR-Cas9 system[J]. Nature Communications, 2017, 8(1):1375. ↩︎
-
Stephenson A , Raper A T , Suo Z . Bidirectional Degradation of DNA Cleavage Products Catalyzed by CRISPR/Cas9[J]. Journal of the American Chemical Society, 2018, 140(10):3743. ↩︎
-
Kuscu, C., Arslan, S., Singh, R., Thorpe, J. & Adli, M. Genome-wide analysis reveals characteristics of off-target sites bound by the Cas9 endonuclease. Nat Biotechnol 32, 677-683, doi:10.1038/nbt.2916 (2014) ↩︎
-
Slaymaker, l. M. et al. Rationally engineered Cas9 nucleases with improved specificity. Science 351, 84-88, doi:10.1126/science.aad5227 (2016) ↩︎